基于Nvidia Clara Parabricks的基因测序加速
更新时间:2025-10-27
概览
Clara Parabricks是由NVIDIA开发,基于GPU的基因组分析软件,包含比对、预处理、突变检测、 UMI、BAM2FQ等多种功能。端到端处理流程包含DNA Germline、DNA Somatic、RNA数据处理。NVIDIA将行业标准版的CPU bwa、gatk等软件进行并行改写,即为Clara Parabricks。对比传统计算模式,在GPU使用Clara Parabricks进行基因分析能够保证一致性的结果,以及更高效的计算。
需求场景
- 基因测序。
- 药物研发。
前提条件
配置步骤
申请Clara Parabricks试用license
- 登录申请页面,填写Clara Parabricks试用申请表,1-3个工作日后会收到一封来自Nvidia邮件,点击邮件中的accept invitation and sign in,登录NGC。
- 登录时,选择external-parabricks-trial-users作为您的组织, 登录后可以看到下图的页面。在左侧目录选择Resources并下载Clara Parabricks license及安装包。
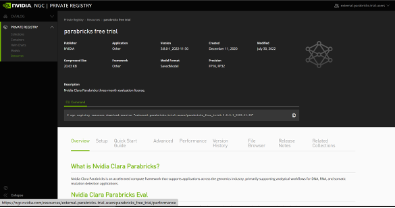
使用Clara Parabricks实现实现DNA及RNA分析流程
下面列举常用功能进行使用说明,详细使用指南请参阅Clara Parabricks User Guide。
fq2bam可实现序列比对,将测序仪产生的fq数据与标准基因组进行比对。此案例中人类标准基因组数据可从NCBI下载,测试数据使用标准样品NA12878,30X WGS,可以从这里下载。
Plain Text
1pbrun fq2bam --ref Ref/Homo_sapiens_assembly38.fasta \
2--in-fq Data/sample_1.fq.gz Data/sample_2.fq.gz \
3--out-bam mark_dups_gpu.bam \
4--out-recal-file recal_gpu.txt \
5--tmp-dir /raid/myrun
6#可按需替换输入fq文件,指定output bam文件名及经过Base Quality校准过后的report文件名Variant caller–Haplotypecaller基于比对产生的bam文件及校正报告,使用gtakHaplotypecaller模块检测生殖系突变。

Plain Text
1pbrun haplotypecaller --ref Ref/Homo_sapiens_assembly38.fasta \
2--in-bam mark_dups_gpu.bam \
3--in-recal-file recal_gpu.txt \
4--out-variants result.vcf
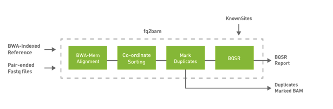
5#可按需替换mark_dups_gpu.bam 及recal_gpu.txt,并指定输出vcf文件名及路径Variant caller-Mutectcaller基于比对产生的bam文件和校正报告,使用gatk Mutechcaller模块,检测体细胞突变。与生殖系突变检测不同,体细胞突变通常是由于后天因素发生。在检测时,一般需要癌组织与正常组织进行比较。
Plain Text
1pbrun mutectcaller \
2--ref Ref/Homo_sapiens_assembly38.fasta \
3--tumor-name tumor \
4--in-tumor-bam tumor.bam \
5--in-normal-bam normal.bam \
6--normal-name normal \
7--out-vcf output.vcf
8#可按需替换肿瘤及正常组织的bam并输出需要指定vcf的文件名及路径。RNA Pipeline比对及检测RNA fusion。
Plain Text
1pbrun rna_fq2bam \
2--in-fq sample_X_1.fq.gz sample_X_2.fq.gz \
3--genome-lib-dir HG38 \
4--output-dir sample_X/ \
5--ref ref.fasta
6可按需替换测序仪产生的fq文件,并指定输出文件目录,此案例中指定名字为“sample_x.out.junction”的文件作为下一步的输入。
7
8pbrun starfusion \
9--chimeric-junction sample_x.out.junction \
10 --genome-lib-dir HG38 \
11 --output-dir sample_X/相关产品
GPU云服务器